神戸大学大学院医学研究科の梶本 武利 (かじもと たけとし) 助教、中村 俊一 (なかむら しゅんいち) 教授らの研究グループは、カリフォルニア大学サンディエゴ校のアレキサンドラ ニュートン教授、アンドリュー マキャモン教授らとの共同研究により、がん細胞が飢餓などのストレスによる死 (アポトーシス※1) を回避する際のブレーキ役として、スフィンゴシン1-リン酸 (S1P)※2 とプロテインキナーゼC (PKC)※3 の直接作用による細胞内シグナリング※4 を、新たに開発した分子センサーと立体構造シミュレーションにより発見しました。本成果により細胞死のブレーキ役が分子の「形」のレベルで明らかになったことで、今後この分子構造情報を基にがん細胞死のブレーキを解除する新たな分子標的薬※5 の探索?設計が可能になると期待されます。
本研究成果は、米国東部時間2019年1月1日午後2時に、国際学術誌「Science Signaling」のオンライン版に掲載されました。

S1P-aPKCシグナリング活性を持つがん細胞は飢餓ストレスの環境下でも死を回避するが、S1P-aPKCシグナリング活性を奪われたがん細胞はアポトーシスを起こして自ら死んでいく。
ポイント
- がん細胞が死 (アポトーシス) を回避するブレーキ役として「S1P-aPKCシグナリング」をaPKCの細胞内活性をモニターする新規分子センサーを用いて新たに発見
- 立体構造シミュレーションとの融合によりS1P-aPKC結合の分子構造情報の取得に成功
- がん細胞が持つ死に対するブレーキを解除する新たな分子標的薬の開発に期待
研究の背景
我々の身体の細胞では、日々様々な要因によってそのDNAが傷つけられています。その多くは修復されますが、一部の細胞では失敗し、これらDNAの修復に失敗した細胞は細胞の自殺とも呼ばれるプログラムされた細胞死 (アポトーシスなど) によって、周辺の正常な細胞に影響を与えることなく自ら死んでいきます。ところががん細胞は様々な方法で死を回避する能力を獲得しており、多くの細胞が死に至る飢餓などのストレス環境下でも生き続けることができます。がんの治療に用いられる多くの抗がん剤はがん細胞のDNAをさらに損傷させてアポトーシスを起こさせるものが一般的ですが、様々なアポトーシス回避能力を備えるがん細胞の全てをアポトーシスへ導くことは難しく、治療抵抗性の一因となっていました。がん細胞が死を回避するブレーキ役を制御することができれば、選択的で副作用の少ない新たながん治療ターゲットになることが期待されており、細胞死回避のメカニズムを明らかにする研究が大変注目されています。ただ、細胞死回避をターゲットとするがん治療薬の開発は未だ実現していません。そこで本研究グループは、創薬に直結する分子の立体構造情報の取得も含めた、新たな細胞死回避メカニズムの解明を目指しました。
研究の内容

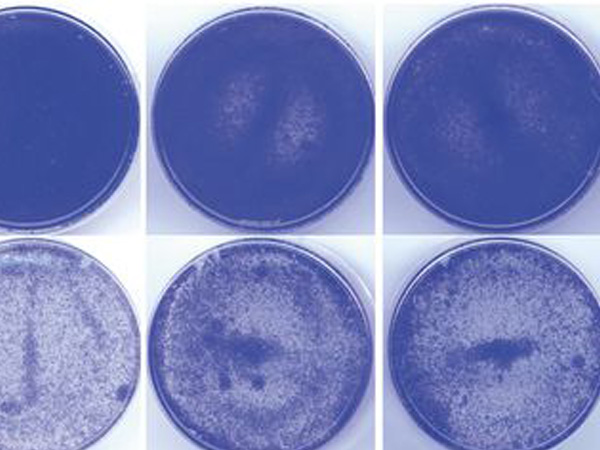
上皮系の癌細胞に、阻害剤を用いてaPKCの働きを抑える処置をすると、アポトーシスの指標となるシグナルが細胞表面に露出する (アポトーシスを起こした細胞が緑色に染まっている)。
がん細胞死のブレーキ役はがん細胞を生かす分子群の中に存在することが予想されました。そこで本研究グループは、がん細胞の生存に関わることが知られている細胞内シグナルタンパク質である非典型プロテインキナーゼC (aPKC) に着目しました。まず上皮癌由来のがん細胞を用いてアポトーシスの有無を観察したところ、aPKCの働きを抑えることでアポトーシスが誘導されたことから (図1)、aPKCはがん細胞の死 (アポトーシス) のブレーキ役として働いていることがわかりました。
次にがん細胞死のブレーキ役としてのaPKCの活性制御メカニズムを明らかにするために、緑色蛍光タンパク質※6 を用いてaPKCに選択的な分子センサー (aPKCセンサー) を開発し、生きた細胞でaPKCの働きを観察できる実験系を構築しました (図2A)。このaPKCセンサーを用いて、がん細胞における恒常的なaPKC活性を引き起こす因子を探索したところ、スフィンゴシン1-リン酸 (S1P) がaPKCに直接結合して活性化させることがわかりました (図2B)。S1Pは多くのがんにおいてその産生量が増加していることが知られています。

A) aPKCセンサーの模式図を示す。aPKCセンサーは、緑色蛍光タンパク質の改変型である水色蛍光タンパク質と黄色蛍光タンパク質間の蛍光共鳴エネルギー移動 (FRET) 現象を用いた1分子FRETセンサーである。① 細胞内でaPKCが活性化し、② aPKCに特異的な基質配列 (赤色部分) がリン酸化 (P) を受けると、③ リン酸化された基質配列が青色の部分に結合して形が大きく変化し、④ 結果FRET現象が起こらなくなるため蛍光が黄色から水色に変わる。このようにして色の変化によりaPKCの活性化状態を生きた細胞の中でモニターすることができる。
B) 上皮系のがん細胞に、S1Pの産生を抑えることで細胞内S1Pを枯渇させる処置をすると、がん細胞内のaPKCの恒常活性が約70%抑えられる (青→赤)。さらに細胞内にS1Pを添加するとaPKC活性が処置なしのレベルまで回復する (赤→黄)。

銀: aPKCの立体構造の一部、赤: aPKCに結合するS1P、青: S1Pの結合に重要なaPKC上のアミノ酸、緑: S1P-aPKC間の水素結合
さらにS1PとaPKCの結合様式を分子構造情報として明らかにするために、タンパク質立体構造データベース※7 上のaPKCの結晶構造を用いてS1Pとの誘導適合ドッキングシミュレーション解析※8 を行ったところ、aPKC上の正の電荷を持つアミノ酸※9 がS1Pの結合に必要であることが予測されました (図3)。またドッキングシミュレーションによって予測されたアミノ酸の機能を失くしたaPKC変異体を遺伝子操作※10 により作製し、aPKCセンサーを用いて細胞内でのaPKC活性を観察したところ、S1Pによる活性化のみが消失していました。またS1Pの制御を受けないaPKC変異体は、飢餓ストレス環境下においてがん細胞をアポトーシスから回避する能力が失われていました。
以上の結果から、正の電荷を持つアミノ酸を介するS1Pの直接作用によるaPKCの恒常的な活性化 (S1P-aPKCシグナリング) が、がん細胞を死 (アポトーシス) から回避するブレーキ役として働いていることがわかりました。
今後の展開
本研究では、がん細胞アポトーシスのブレーキ役として、S1Pの直接作用によるaPKCの恒常的な活性化シグナリングを新たに発見しました。本研究成果の重要なポイントは、S1PとaPKCの結合情報を分子構造レベルで取得することに成功したことです。今後は、今回明らかになった「がん細胞アポトーシスのブレーキ役としてのS1P-aPKCシグナリング」の組織?生体レベルでの検証を進め、最終的には「S1P-aPKC結合の分子構造情報」を基に、がん細胞アポトーシスのブレーキ解除をターゲットとする新たな分子標的薬開発への早期展開が期待できます。
用語解説
- ※1 アポトーシス
- 細胞が自己の持つプログラムにより計画的に脱落死する現象。細胞の自殺とも呼ばれる。DNAの損傷や栄養飢餓などにより誘導される。また生体の発生において、例えば人の指などの形成過程にも働く。
- ※2 スフィンゴシン1-リン酸 (Sphingosine 1-phosphate (S1P))
- 生体の細胞膜を構成するスフィンゴ脂質 (スフィンゴミエリンなど) の代謝産物。セラミド、スフィンゴシンを経て、スフィンゴシンキナーゼによってリン酸化を受けることで産生する。細胞内外でシグナル分子として働く。
- ※3 プロテインキナーゼC (Protein Kinase C (PKC))
- セリン?スレオニンリン酸化酵素の一種。在来型PKC (conventional:α、βI、βII、γ )(cPKC)、新型PKC (novel:δ、ε、η、θ)(nPKC)、非典型PKC (atypical:ζ、λ/ι)(aPKC) の3サブファミリー、10種に分類される。細胞内シグナリングの鍵となるタンパク質であり、がんや神経変性疾患などの原因となる。
- ※4 細胞内シグナリング
- 細胞内での分子同士の情報 (信号) の流れ。タンパク質とタンパク質の結合、低分子とタンパク質の結合、タンパク質のリン酸化修飾などにより、構造変化を起こさせるなどして情報 (信号) を伝える。1つの分子が多数の分子へ信号を渡し、ソーシャル?ネットワーキング?サービス (SNS) のように情報を拡散させることで、微弱なシグナルを大きなシグナルへと増幅する働きがある。
- ※5 分子標的薬
- 主にがんの領域で用いられ、特定の分子を選択的に制御する機能を持ち、がん細胞のみを狙い撃ちにする医薬品。従来品よりも副作用が少なく効果が高い。
- ※6 緑色蛍光タンパク質 (Green Fluorescence Protein (GFP))
- オワンクラゲの発光の原理となる緑色の蛍光性を持つタンパク質。青色の蛍光を吸収することで励起状態となり、基底状態に戻る際に緑色の蛍光を発する。遺伝子操作により水色、黄色など様々な改変型が開発されており、生きた細胞内でのタンパク質の蛍光標識などに用いられる。2008年ノーベル化学賞。
- ※7 タンパク質立体構造データベース
- タンパク質の立体構造データを登録しているデータベース。登録されている立体構造データは、主にX線結晶解析、核磁気共鳴解析やクライオ電子顕微鏡解析などによって同定されている。主なものに国際的な公共のデータベースであるProtein Data Bank (PDB) がある。
- ※8 誘導適合ドッキングシミュレーション
- 低分子-タンパク質間相互作用における複合体の安定構造をコンピュータ上で計算的に推定する手法がドッキングシミュレーションである。通常のドッキングシミュレーションでは、タンパク質の構造を剛体として固定し、低分子のみに柔軟性を持たせて計算を行う。誘導適合ドッキングシミュレーションでは、低分子とタンパク質の両方に柔軟性を持たせ、お互いが結合に適した形状へと誘導適合することで真に正しいドッキング計算が行われるため、高精度な分子構造情報の取得が可能となる。
- ※9 アミノ酸
- 20種類のアミノ酸が存在し、遺伝子の設計図通りに連結されることでタンパク質を構成する。荷電アミノ酸の分類では、正の電荷を持つアミノ酸 (アルギニン、リジン、ヒスチジン)、負の電荷を持つアミノ酸 (アスパラギン酸、グルタミン酸)、その他電荷を持たないアミノ酸に分類される。
- ※10 遺伝子操作
- 遺伝子を、1塩基のレベルで他の塩基へ変異させたり、ある遺伝子領域を欠失させたり、他の遺伝子とつなげたりして、人為的に操作すること。
謝辞
本研究は、文部科学省、アメリカ国立衛生研究所、神戸大学、カリフォルニア大学サンディエゴ校、および中谷医工計測技術振興財団の支援を受けて、文部科学省が推進する国際共同研究強化事業および神戸大学-カリフォルニア大学サンディエゴ校間の学術交流協定の一環として行われました。
論文情報
- タイトル
- “Activation of Atypical Protein Kinase C by Sphingosine 1-Phosphate Revealed by Atypical Protein Kinase C-Specific Activity Reporter”
- DOI
- 10.1126/scisignal.aat6662
- 著者
- Taketoshi Kajimoto, Alisha D. Caliman, Irene S. Tobias, Taro Okada, Caila A. Pilo, An-Angela Van, J. Andrew McCammon, Shun-ichi Nakamura, Alexandra C. Newton
- 掲載誌
- Science Signaling