神戸大学の坂田雅之学術研究員、源利文教授らと、長浜バイオ大学の倉林敦准教授ら、いであ株式会社の中村匡聡ら、京都大学の西川完途准教授など7つの機関からなる国際共同研究グループは、両生類(カエル類?イモリ類?サンショウウオ類)の体表の粘液や糞などとともに水中に放出された微量なDNA(環境DNA)を分析可能な増幅してまとめて分析することによって、棲んでいる両生類の種類を判定する技術を開発しました。この技術は捕獲や目視などこれまで調査者の専門的知識や天候?季節などに結果が左右されがちだった従来法に対し、水を汲むだけで誰でもそこにすむ両生類を調べることができる画期的な手法となることが期待されます。本研究成果は、2月21日(月)に、オンライン国際学術誌「Metabarcoding and Metagenomics」に掲載されました。
ポイント
- 生物多様性のモニタリングは、自然生態系全般の保全に重要ですが、特に個体数の減少が顕著な両生類については、その重要性が増しています。
- しかし、両生類は、主に夜行性であること、幼生(オタマジャクシ)と成体では異なる環境に生息すること、個体の採捕や種の同定には専門的な知識が必要なことなどから、調査ごとに結果のばらつきが生じ、統一規格での正確なモニタリングが難しいことが課題でした。
- 近年、環境中に放出された生物外DNA(環境DNA)を解析する環境DNA分析手法を用いて生物のモニタリングが行われるようになっており、特に魚類では手法が確立されているため、一般的になりつつあります。一方で、両生類においては標準的な分析手法が確立されておらず、発展途上であったため、両生類の環境DNAをまとめて分析し、調査地点にどのような種が生息しているのかを調べるための分析手法を開発しました。
- まず、両生類を対象とした環境DNA分析の分析手法を複数設計し、性能を事前評価しました。その後、そのうちの一つの手法を用いて、日本中の10の農業地域122地点において、環境DNA分析と従来法による調査(網を用いた採捕調査や目視調査)を同時に行い、検出された両生類を比較しました。
- 新たに作成した分析手法では、両生類に分類される3目(有尾目?無尾目?無足目)を全て検出可能であることが示されました。また、野外調査の結果、すべての地域で従来法による調査より今回開発した環境DNA分析の方が多くの種を検出することができ、この手法の有効性が示されました。
研究の背景
両生類の多様性の減少は世界的に進行しており、その保全のためには生息地などの基本情報を収集するためのモニタリングが重要です。従来の両生類のモニタリングでは、目視調査や鳴き声調査、採捕調査が行われてきましたが、両生類は、個体サイズが小さいこと、主に夜行性であること、調査効率が天候や季節に大きく左右されること、種の判別に専門的な知識が必要なことなどから、広範囲を統一的にモニタリングし、生息状況を評価することが困難でした。この10年程度の間に、生き物の糞や粘液と共に環境中に放出された生体外のDNA(環境DNA)を分析し、生き物の分布を調べる環境DNA分析技術が大きく発展してきました。この技術は、基本的に調査地で水を汲んでそこに含まれる環境DNAを分析することにより、どのような生き物が生息しているかなどを知ることができるものであり、従来法を補完しうるモニタリング手法として、近年、注目されています。特に魚類においては、他の生物群に先行して標準的な分析手法が確立しているため、環境DNA分析を用いた生物多様性のモニタリングが一般的になりつつあります。一方で、両生類を対象とした環境DNA分析での生物モニタリングは、いまだ発展途上であると言えます。その理由の一つとして、環境DNA分析には、対象種や対象分類群に合わせた分析手法の開発が必要であり、両生類を網羅的に検出するための分析手法の開発?実用化に課題が残されていることが挙げられます。環境DNA分析で両生類を網羅的に検出することのできる分析手法が開発されれば、種判別のための専門的な知識や調査経験がなくとも両生類のモニタリングが可能になる上、全国規模のような広範囲のモニタリング調査などにおいて、統一規格での調査が可能になることが期待されます。本研究では、両生類調査のための分析手法の開発とモニタリングにおける評価を行うことで、魚類で行われているように、両生類においても生物モニタリングに環境DNA分析が一般的に使用されるようになるための大きな基盤となると考えられます。
研究の内容
両生類を対象としたユニバーサルプライマーの設計と評価
インターネット上のオープンデータベースから合計1,034の両生類のDNA配列を取得し、種間で同じ塩基配列を持っている領域を探し出し、両生類のDNAを網羅的に増幅できるような分析系をミトコンドリアの16SrRNA領域に5つ設計しました。その後、それぞれのセット間で、両生類のDNAの増幅しやすさ、両生類以外のDNAを増やしてしまう可能性(特異性)、両生類の種をきちんと判別できるか(種解像度)などの項目をコンピューター上で検討しました。また、両生類の個体から得たDNAを用いてきちんと色々な種類の両生類のDNAを増幅することができるかを実験的に確認しました。その結果、どの分析系もきちんと両生類のDNAを増幅することができました。これらの事前検討の結果から、特異性と種解像度が秀でている1つの分析系を選択し、野外調査でモニタリングにおける有効性を評価することにしました。
野外調査における従来調査と比較した有効性の検証
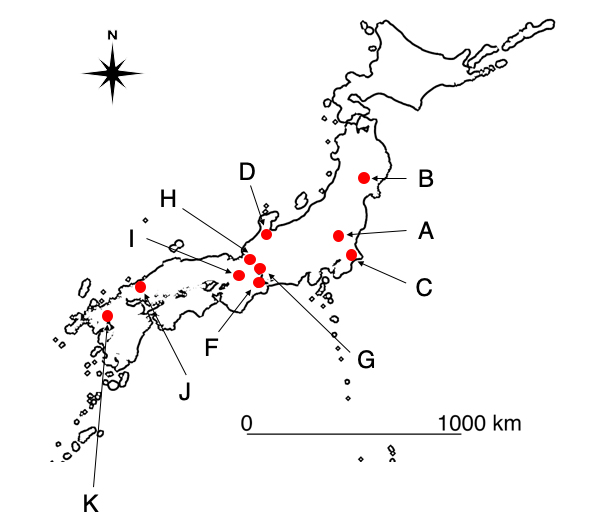
設計した分析系の野外モニタリングにおける有効性を確認するため、2019年の6~9月に日本の農業生態系(水田生態系)10地域122地点において、野外調査を行いました(図1)。野外調査では、各地域のため池や農業用水路で環境DNA分析と従来法による調査(採捕?目視調査)の両方を実施し、検出された両生類の種数や種組成を比較しました。野外調査の結果、図2に示すようにどの地域においても検出種数は環境DNA分析の方が通常の調査を上回りました。したがって、本研究で開発された両生類を対象とした分析系を用いた環境DNA分析は、野外モニタリングにおいても有効であることが示されました。

今後の展開
- 本研究で開発した分析手法は、両生類のDNAを網羅的に検出できるため、これを用いた環境DNA分析でのモニタリングは、希少種や個体数の少ない種の迅速な分布調査に対しても貢献することが期待されます。
- 環境DNA分析手法は、捕獲や目視などの従来の調査手法よりも季節や天候などといった環境要因の影響を受けにくいため、安定して両生類を検出することが可能であり、全国規模のような広範囲の調査に対して、統一した調査手法を提供できると考えられます。
- 本技術を有効に活用していくためには、増幅したDNAがどの種由来のDNAであるかを判別するための参照データの蓄積が望まれます。参照データがより蓄積されることで、より多様な国々?地域でこの技術が利用可能になり、両生類のモニタリングに大きく貢献することが期待されます。
用語解説
- 環境DNA(eDNA)
- 水中や土壌などの環境中に存在する生物由来のDNA。PCRを用いて環境DNAを分析することで、現在ないし過去にそこに生息していた生物種の網羅的な同定、標的種の生息状況の推定?把握ができる。環境DNA調査は、保全生物学、生態学、系統分類学、微生物学、古生物学などの広範な分野の研究に利用されている。魚類では環境DNA分析を用いた研究が先行しており、生物モニタリングにおいても一般的になりつつある。一方で、両生類においては、十分に多様性調査に利用可能な検出系が確立されていないこともあり、発展途上である。
- (環境DNA)メタバーコーディング
- 複数種をまとめて検出する手法。対象分類群に共通する塩基配列領域に設計されたユニバーサルプライマーと呼ばれるプライマーを用いることで、対象分類群に含まれる複数種のDNAをまとめてPCRで増幅する。その後、増幅産物を超並列シークエンスで網羅的に配列決定し、データベースに登録されている配列と照合されることで種が推定される。
論文情報
- タイトル
- “Development and evaluation of PCR primers for environmental DNA (eDNA) metabarcoding of Amphibia”
- DOI
- 10.3897/mbmg.6.76534
- 著者
- Masayuki K. Sakata, Mone U. Kawata, Atsushi Kurabayashi, Takaki Kurita, Masatoshi Nakamura, Tomoyasu Shirako, Ryosuke Kakehashi, Kanto Nishikawa, Mohamad Yazid Hossman, Takashi Nishijima, Junichi Kabamoto, Masaki Miya, Toshifumi Minamoto
- 掲載誌
- Metabarcoding and Metagenomics