広島修道大学の岡西政典 助教、東京大学大学院理学系研究科附属臨海実験所、神戸大学、(株) 環境総合リサーチ、京都大学からなる研究グループが、海中の無脊椎動物の体表の粘液や排泄物から水中に放出されたDNA (環境DNA) を分析することで、そのDNAの持ち主の種類を判別する技術を開発しました。
海洋は地球の面積の70%を占めており、そこにすむ生物は我々に大きな恩恵をもたらしています。しかし、海洋環境推定のために必要な生物の種類調査には、潜水や網を用いた作業が必要で、大きな労力と時間がかかっていました。また海洋の大部分を占める深海においてはこの作業がさらに困難でした。
今回の研究では、ヒトデの仲間であるクモヒトデをターゲットとして、それらが水中に放出した環境DNAを調べることで、その種類を判別する技術を開発しました。「メタバーコーディング」と呼ばれるこの技術の研究例は、日本の海では魚類や甲殻類でのみ知られており、深海域での研究例もありませんでした。
この技術によって、魚類や甲殻類などの移動力の高い生物だけでなく、移動性が低くその環境から動かない生物も対象にすることで、より詳しく海洋環境を推定できるようになると期待できます。また今後ウニやナマコなどの他の生物の調査に応用されれば、アクセスが難しい深海域での生物のモニタリングを、誰でも、広範囲にわたり、短時間で行うことが可能になります。
本研究成果は2023年2月28日にブルガリアの科学雑誌『Metabarcoding & Metagenomics』で公開されました。
ポイント
- 従来、海洋生物の種類調査には多大な費用と労力がかかっていた
- 海水中に漂っているDNAからヒトデの仲間の種類を判定できる技術を開発した
- 飼育水槽と、相模湾の水深270mまででその検出力を検証した
- 今後無脊椎動物を含むさまざまな生物を用いた環境モニタリングが可能となる
- クモヒトデ類においては世界初、深海域においては日本初の技術開発となる

研究の背景と経緯
海洋は地球の面積の70%を占め、我々の生活と密接に関わっています。例えば世界人口の1/3が集中する沿岸域では人口増加と経済発展が著しく、沿岸生態系の経済的な価値は生物資源由来で約2,500兆円と見積もられています。一方でこのまま海洋環境汚染が進めば、2050年までに約1,820兆円分が失われると考えられています。この損失を防ぐために、生物資源の保全は緊急に解決すべき課題です。
そのためには、各地、各年代で海洋生物の種数を比較し、その推移を把握することは不可欠です。しかし海洋調査には多大な労力がかかり、漁業関係者との調整も必要なことから、そのような調査がなかなか進んでいませんでした。
このような状況の中「環境DNAメタバーコーディング法」がこの問題解決の糸口を提供しつつあります。これは水中に残されたDNA (環境DNA) から、簡便かつ迅速に種数を把握する技術です。国内でもいくつかの海洋生物で成果が上げられており、その海域に生息する生物種数を、バケツ一杯の水を汲むだけで検出することに成功しています。
これらの解析には、①DNAを多く海水中に放出する生物であること、②浅海から深海まで幅広く生息し、個体数が多いこと、③予め種がわかっているDNA配列情報 (リファレンス配列) が登録されていること、といった条件が必要です。その条件を満たす海洋生物として、これまではプランクトンが解析の対象とされてきました。一方で、海底で暮らす「底生生物」の調査も欠かせません。これまで底生生物では魚類や甲殻類での研究が知られていましたが、海底環境のモニタリングにおいては、それらに加え、移動力に乏しい (その環境からあまり動かない) 生物の情報も併せることで、より詳細に環境を推定できると考えられます。そこで本研究では、ウニ?ヒトデ?ナマコの仲間であり、あらゆる海底環境に群生しており、これらの条件を満たしうる「クモヒトデ類」に着目しました。この生物について、世界で初めてとなる「クモヒトデメタバーコーディグ」技術を開発し、その多様性が高い相模湾においてその性能を検証しました。
研究の成果

A. 実験所桟橋での採水、B. C. 実験所近傍の諸磯浅海でのスキューバ採水、D. 深海域での採水、E. 実験所の飼育水槽、F. 個別の飼育水槽
クモヒトデは、概して目に見えるサイズで個体数が多く、さまざまな海底環境に生息していることから、環境DNAを豊富に放出すると考えられる海産無脊椎動物です (図1)。ヒトデやウニが所属する棘皮動物の中では最も種類が多く (約2100種)、砂泥、岩場、海山など、種ごとに生息環境が決まっているため、海底環境の指標生物や環境DNAメタバーコーディングの対象生物として有望です。
そこで本研究ではまず、国際的なDNAデータベース (INSD) に登録されていたクモヒトデのDNA配列のうち、様々なグループに所属するクモヒトデ132種の配列を取得しました。ここから、クモヒトデの種の違いを表すと考えられる遺伝子領域を挟み込みようにDNAプライマー (注1) を設計しました。この遺伝子領域は、海水中で劣化し、短く分断されたDNAも検出できる程度の長さ (約130bpと約180bp (注2) の二領域) に設計しています。
また、メタバーコーディングでは、環境から得られた多数のDNA配列がどの生物に由来するのかを決定するための「参照DNA配列」が必要不可欠です。INSDに登録されたクモヒトデ配列には由来が不明瞭で、参照配列に用いることができないものも含まれていました。そこで本研究では、相模湾で10年以上にわたって蓄積されたクモヒトデ類標本を同定 (注3) し、うち60種に基づく参照DNA配列データベースを構築しました。
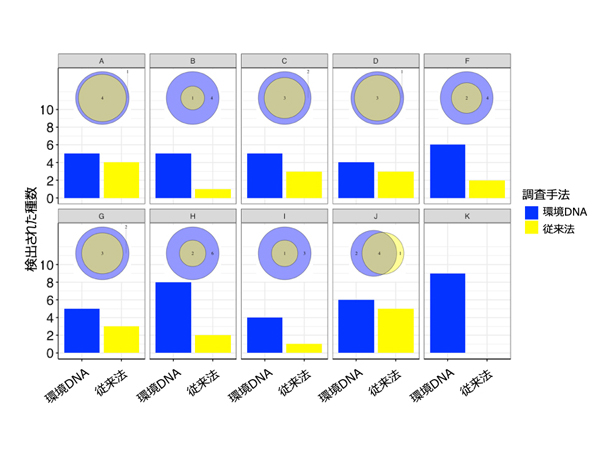
このプライマーの性能を検証するために、東京大学大学院附属臨海実験所のクモヒトデ飼育水槽水 (合計1リットル) から抽出した環境DNAを用いてプライマーの性能を検証しました。その結果、12飼育種中11種の検出に成功しました。また実験所が臨む相模湾の表層水、水深2m~270mの環境DNAを抽出し、同様に実験を行ったところ、現地に生息する45種のうち27種を検出しました (図2)。さらに、クモヒトデだけでなく、浅海に生息するヒトデ、ウニ、ナマコ、ウミシダなどの棘皮動物門 (注4) の他、貝類、甲殻類、コケムシ類、ヒモムシ類などの海産無脊椎動物の検出も確認されました。
今後の展開
海底環境のモニタリングにおいては、魚類や甲殻類に加え、移動力に乏しい (その環境からあまり動かない) 生物の情報も併せることで、より詳細に環境を推定できると考えられます。そのような生物に、簡便なメタバーコーディング法が用いられなかった理由の一つに、参照DNA配列が充実していなかったという点が挙げられます。
この問題解決のためには、その生物が明瞭に分類されることが肝要です。本研究では、クモヒトデという、メタバーコーディングに有用な生物に着目し、種の分類を明らかにした上で参照DNA配列を構築したことにより、新たな技術の開発に成功しました。
今後、他の海洋生物でも分類が進み、プライマーの開発と参照DNA配列の構築によってメタバーコーディングが可能になれば、これまでに考えられなかった精度での海洋環境モニタリングが可能になると考えられます。特に、採水だけで調査ができるという点は、生物の種類が多いにも関わらず、漁業権の問題で曳網が難しい深海底での調査が可能になる点で画期的です。
また、今回は生息記録があるにもかからず検出できなかった種も見られました。これが実験における問題か、野外でのDNAの分解の問題かを確かめるため、今後は日本近海を中心に採水を行い、今回開発した手法の精度検証と改善を行っていこうと考えています。
用語解説
- 注1) DNAプライマー
- ポリメラーゼ連鎖反応 (PCR) で用いられる一本鎖DNAのこと。PCRで増幅したいDNA (テンプレート) の両脇に設計され、それらがテンプレートと結合することにより、そこを起点とした合成反応が始まる。
- 注2) 約130bpと約180bp
- base pair (塩基対) の略。DNAの構成要素の4つの塩基が対になった状態を表す。基本的にはこのbpの何億もの並びが生物の特徴を決めている。
- 注3) 同定
- 生物の名前を調べること。
- 注4) 棘皮動物門
- ウニ、ヒトデ、ナマコ、クモヒトデ、ウミユリなどの動物を含むグループ。ほぼ全て海産で、五放射相称 (星形) の体や、炭酸カルシウム性の骨片を持つという共通点を持つ。
研究助成
- 研究課題名:「棘皮動物門クモヒトデ綱の科階級群における分子系統解析と腕の骨格の進化」
研究種目:科研費基盤C
研究課題番号:25440226 - 研究課題名:「博物館標本DNAに基づく海産無脊椎動物ニシキクモヒトデの保全学的研究」
研究種目:科研費基盤C
研究課題番号:21K05632 - 研究課題名:「新たな海洋保護区(沖合海底自然環境保全地域)管理のための深海を対象とした生物多様性モニタリング技術開発」
研究種目:環境研究総合推進費
研究課題番号:JPMEERF20S20704
論文情報
- タイトル
- “Development of two new sets of PCR primers for eDNA metabarcoding of brittle stars (Echinodermata, Ophiuroidea)”
- DOI
- 10.3897/mbmg.7.94298
- 著者
- Masanori Okanishi, Hisanori Kohtsuka, Qianqian Wu, Jumpei Shinji, Naoki Shibata, Takashi Tamada, Tomoyuki Nakano, Toshifumi Minamoto
- 掲載誌
- Metabarcoding and Metagenomics